Ученые NVIDIA и Гарвардского университета совершили прорыв в области геномики
Большинство клеток тела несут с собой полную копию ДНК человека, с миллиардами базовых пар, сжатых в ядре. Но отдельная клетка использует только тот подраздел генетической информации, который ей необходим для функционирования, при этом каждый тип клетки - клетки печени, крови или кожи - активируют различные гены. Области ДНК, которые определяют уникальную функцию клетки, открываются для легкого доступа, в то время как остальное остается незадействованным.
Исследователи из NVIDIA и Гарвардского университета разработали инструментарий на базе технологии глубокого обучения, который поможет ученым исследовать эти доступные области ДНК, даже когда данные образцов зашумлены или ограниченные - что часто происходит при раннем выявлении рака и других генетических заболеваний.
Система AtacWorks, описание которой было опубликовано в журнале Nature Communications, способна распознавать данные секвенирования, так и определять области с доступной ДНК, и может выполнить анализ всего генома всего за полчаса с помощью графических процессоров NVIDIA Tensor Core. Система доступна на NGC - концентраторе программного обеспечения NVIDIA, оптимизированного для графического процессора (GPU) этой фирмы.
AtacWorks работает с ATAC-seq, популярным методом поиска открытых участков генома как в здоровых, так и в больных клетках, что позволяет получить критическую информацию для разработки соответствующего лекарства.
ATAC-seq обычно требует, чтобы десятки тысяч клеток получали чистый сигнал, что очень затрудняет исследование редких типов клеток, таких как стволовые клетки, которые продуцируют клетки крови и тромбоциты. Применяя AtacWorks к данным ATAC-seq, можно достичь такого же качества результатов всего лишь с десятками клеток, что позволит ученым узнать больше о последовательностях, активных в редких типах клеток, и выявить мутации, которые делают людей более уязвимыми к болезням.
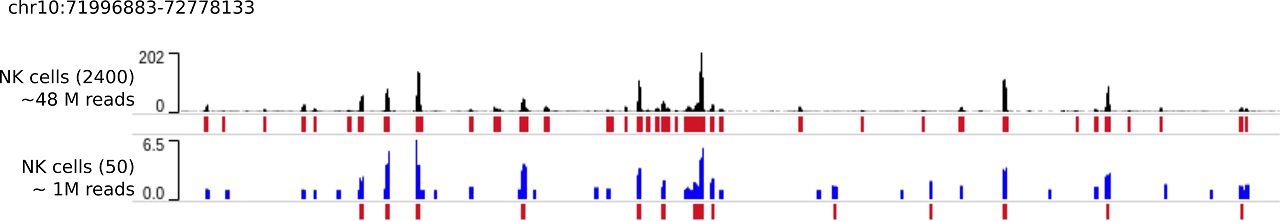
"С помощью AtacWorks мы можем проводить эксперименты с одиночными клетками, для которых обычно требуется в 10 раз больше клеток", - говорит соавтор статьи Джейсон Буэнростро, доцент Гарвардского университета и разработчик метода ATAC-seq.
ATAC-seq разработан в 2013 году как способ сканирования эпигеномов, чтобы обнаружить места с доступными областями в хромосоме, известные как хроматин. Этот метод измеряет интенсивность сигнала в каждой области генома. Пики сигнала соответствуют участкам с открытой ДНК. Чем меньше клеток доступно, тем шумнее появляются данные, что затрудняет определение того, какие участки ДНК доступны.
Система AtacWorks, базирующаяся на конволюционной нейронной сети, была обучена работе с мечеными парами соответствующих наборов данных ATAC-seq: одним высококачественным и одним шумным. Учитывая уменьшенную копию данных, модель научилась предсказывать точную высококачественную версию и определять пики в сигнале.
Ученые обнаружили, что с помощью AtacWorks они могли проводить исследования с меньшим количеством клеток, что значительно снижало затраты на отбор проб и секвенирование.
Анализ также становится быстрее и дешевле с помощью AtacWorks. При работе на графических процессорах NVIDIA Tensor Core модель заняла менее 30 минут для вывода результатов по всему геному. Это процесс, который занял бы 15 часов в системе с 32 процессорными ядрами.
"При очень редких типах клеток невозможно изучить различия в их ДНК с помощью существующих методов, - сказала исследователь NVIDIA Авантика Лал (Avantika Lal). - AtacWorks может помочь не только снизить стоимость сбора данных о доступности хроматина, но и открыть новые возможности в области обнаружения и диагностики лекарств".